Begjin materiaal: RNA
Kwantitative reverse transcription PCR (RT-qPCR) is in eksperimintele metoade dy't brûkt wurdt yn PCR-eksperiminten mei RNA as útgongsmateriaal.Yn dizze metoade wurdt totaal RNA as messenger RNA (mRNA) earst transkribearre yn komplementêr DNA (cDNA) troch reverse transkriptase.Dêrnei waard in qPCR-reaksje útfierd mei it cDNA as sjabloan.RT-qPCR is brûkt yn in ferskaat oan molekulêre biologyapplikaasjes, ynklusyf analyse fan gen-ekspresje, RNA-ynterferinsje-validaasje, mikroarray-validaasje, patogeendeteksje, genetyske testen, en sykteûndersyk.
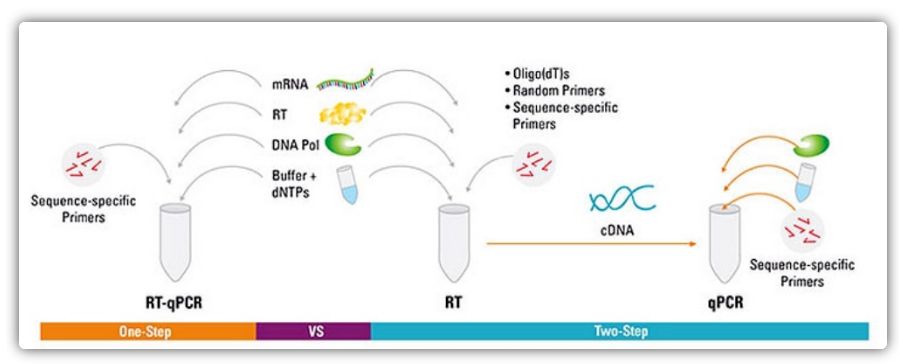
Ien-stap en twa-stap metoaden foar RT-qPCR
RT-qPCR kin wurde berikt troch in ien-stap of twa-stap metoade.Ien-stap RT-qPCR kombinearret reverse transkripsje en PCR amplification, wêrtroch reverse transcriptase en DNA polymerase te foltôgjen de reaksje yn deselde buis ûnder deselde buffer betingsten.Ien-stap RT-qPCR fereasket allinich it gebrûk fan sekwinsje-spesifike primers.Yn twa-stap RT-qPCR wurde omkearde transkripsje en PCR-amplifikaasje útfierd yn twa buizen, mei ferskate optimalisearre buffers, reaksjebetingsten en strategyen foar primerûntwerp.
| Foardiel | Neidiel | |
| Ien stap | Dizze metoade hat minder eksperimintele flater as beide reaksjes wurde dien yn ien buis
Minder pipetteerstappen ferminderje it risiko op kontaminaasje
Geskikt foar fersterking / screening mei hege trochput, fluch en reprodusearber | Twa-stap reaksjes kinne net wurde optimalisearre apart
Om't de reaksjebetingsten kompromittearre wurde troch it kombinearjen fan de twastapsreaksje, is de gefoelichheid net sa goed as dy fan de twastapmetoade
It oantal doelen ûntdutsen troch ien stekproef is lyts |
| Twa stappen | Mooglikheid om stabile cDNA-biblioteken te meitsjen dy't foar lange perioaden kinne wurde opslein en brûkt yn meardere reaksjes
Doelgenen en referinsjegenen kinne wurde fersterke út deselde cDNA-bibleteek sûnder de needsaak foar meardere cDNA-biblioteken
Reaksjebuffers en reaksjebetingsten dy't optimisaasje fan ienige reaksjerinnen mooglik meitsje
Fleksibele seleksje fan triggerbetingsten | It brûken fan meardere buizen, en mear pipetteerstappen fergruttet it risiko fan DNA-fersmoarging, en tiidslinend.
Fereasket mear optimisaasje dan de ienstapmetoade |
Relatearre produkten:
RT-qPCR Easyᵀᴹ (One Step)-SYBR Green I
RT-qPCR Easyᵀᴹ (ien stap)-Taqman
RT Easyᵀᴹ I Master Premix foar earste-string CDNA-synteze
Real Time PCR Easyᵀᴹ-SYBR Green I Kit
Seleksje fan totale RNA en mRNA
By it ûntwerpen fan in RT-qPCR-eksperimint is it wichtich om te besluten oft jo totaal RNA of suvere mRNA brûke as sjabloan foar omkearde transkripsje.Hoewol mRNA in bytsje hegere gefoelichheid kin leverje, wurdt totale RNA noch hieltyd faak brûkt.De reden hjirfoar is dat totale RNA in wichtiger foardiel hat as útgongsmateriaal as mRNA.Earst, it proses fereasket minder suvering stappen, dat soarget foar bettere kwantitative herstel fan sjabloan en bettere normalization fan resultaten te begjinnen sel nûmers.Twads foarkomt it de mRNA-ferrikingsstap, dy't de mooglikheid fan skewe resultaten kin foarkomme fanwegen ferskate werhellingen fan ferskate mRNA's.Oer it algemien, om't yn 'e measte tapassingen de relative kwantifikaasje fan it doelgen wichtiger is dan de absolute gefoelichheid fan' e deteksje, is totale RNA yn 'e measte gefallen geskikter.
Omkearde transkripsje primer
Yn 'e twa-stapmetoade kinne trije ferskillende metoaden brûkt wurde om de cDNA-reaksje te primen: oligo (dT) primers, willekeurige primers, of sesje-spesifike primers.Typysk wurde oligo (dT) primers en willekeurige primers yn kombinaasje brûkt.Dizze primers annealje oan 'e sjabloan mRNA-string en leverje omkearde transkriptase mei in útgongspunt foar synteze.
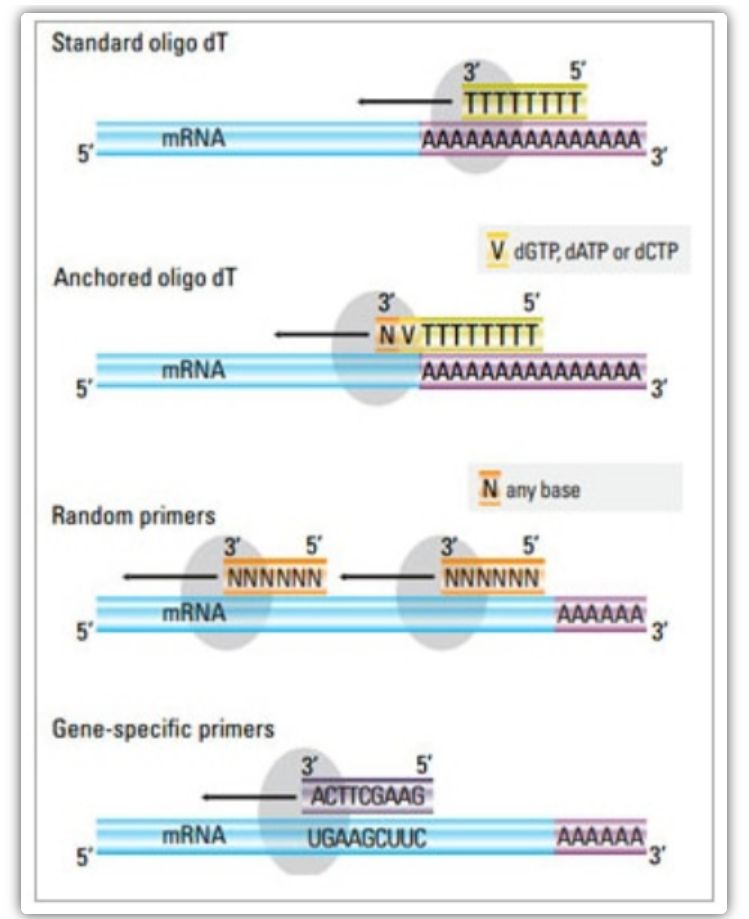
| Primer seleksje | Struktuer en funksje | Foardiel | Neidiel |
| Oligo(dT) primer (of ferankere oligo(dT) primer) | Utwreide annealing nei thymine-residuen by de poly (A) sturt fan mRNA;anker oligo (dT) primer befettet in G, C, of A oan it 3'-ein (ankerside) | Synteze fan cDNA fan folsleine lingte fan poly(A)-tailed mRNA
Tapaslik as minder útgongsmateriaal beskikber is
Anchoring site soarget derfoar dat de oligo (dT) primer bindet oan de 5' poly (A) sturt fan it mRNA | Allinich geskikt foar it fersterkjen fan genen mei poly (A) sturten
Krij cDNA ôfkoarte fan 'e priming site *2 yn poly (A)
Foarindich om te binen oan it 3′ ein *
*Dizze mooglikheid wurdt minimalisearre as ferankere oligo(dT) primers wurde brûkt |
| willekeurige primer
| 6 oant 9 basen yn 'e lingte, dy't annealje kinne oan meardere siden by RNA-transkripsje | Annealje oan alle RNA's (tRNA, rRNA, en mRNA)
Geskikt foar transkripsjes mei wichtige sekundêre struktuer, of as minder útgongsmateriaal beskikber is
Hege cDNA-opbringst | cDNA wurdt omkearde transkribearre fan alle RNA, wat normaal net winske is en it sinjaal fan it doel mRNA kin verdunde
krij ôfkoarte cDNA |
| sequence-spesifike primers | Oanpaste primers rjochte op spesifike mRNA-sekwinsjes | spesifike cDNA bibleteek
Ferbetterje gefoelichheid
Mei help fan reverse qPCR primers | Allinnich beheind ta de synteze fan ien doelgen |
Reverse transkriptase
Omkearde transkriptase is in enzyme dat RNA brûkt om DNA te syntetisearjen.Guon omkearde transkriptases hawwe RNase-aktiviteit en kinne RNA-strengen yn RNA-DNA-hybride stringen nei transkripsje degradearje.As it gjin RNase enzymatyske aktiviteit hat, kin RNaseH wurde tafoege foar hegere qPCR-effisjinsje.Faak brûkte enzymen omfetsje Moloney murine leukemy firus reverse transcriptase en avian myeloblastoma virus reverse transcriptase.Foar RT-qPCR is it ideaal om in omkearde transkriptase te kiezen mei hegere thermostabiliteit, sadat cDNA-synteze kin wurde útfierd by hegere temperatueren, en soarget foar suksesfolle transkripsje fan RNA's mei hegere sekundêre struktuer, wylst se har folsleine aktiviteit yn 'e reaksje behâlde, wat resulteart yn hegere cDNA-opbringsten.
Relatearre produkten:
Foreasy M-MLV Reverse Transcriptase
RNase H-aktiviteit fan reverse transkriptase
RNaseH is yn steat om RNA-strengen te degradearjen fan RNA-DNA-duplexen, wêrtroch effisjinte synteze fan dûbelstrenged DNA mooglik is.By it brûken fan lange mRNA as sjabloan kin it RNA lykwols te betiid ôfbrutsen wurde, wat resulteart yn ôfkoarte cDNA.Dêrom is it faaks foardielich om RNaseH-aktiviteit te minimalisearjen tidens cDNA-kloning as synteze fan lange transkrippen winske is.Yn tsjinstelling binne omkearde transkriptasen mei RNase H-aktiviteit faak foardielich foar qPCR-tapassingen, om't se it smelten fan RNA-DNA-duplexes ferbetterje tidens de earste syklus fan PCR.
Primer ûntwerp
PCR-primers dy't brûkt wurde foar de qPCR-stap yn RT-qPCR moatte ideaal wurde ûntwurpen om in ekson-ekson-junction te spannen, wêr't in amplifikaasjeprimer mooglik in werklike exon-intron-grins kin oerspanje.Sûnt intron-befette genomyske DNA-sekwinsjes wurde net fersterke, ferleget dit ûntwerp it risiko fan falske positiven fersterke troch kontaminearjen fan genomysk DNA.
As primers net kinne wurde ûntworpen om exons of exon-exon-grinzen te skieden, kin it nedich wêze om RNA-samples te behanneljen mei RNase-frije DNase I of dsDNase om genomyske DNA-fersmoarging te ferwiderjen.
RT-qPCR kontrôle
In omkearde transkripsje negative kontrôle (-RT-kontrôle) moat opnommen wurde yn alle RT-qPCR-eksperiminten om DNA-fersmoarging te detektearjen (lykas genomysk DNA of PCR-produkten fan eardere reaksjes).Dizze kontrôle befettet alle reaksjekomponinten útsein reverse transkriptase.Sûnt omkearde transkripsje net foarkomt mei dizze kontrôle, as PCR-amplifikaasje wurdt waarnommen, is fersmoarging fan DNA it meast wierskynlik.
Post tiid: Aug-02-2022








